生体分子を含む様々な分子システムの大規模・長時間分子動力学(MD)シミュレーションをGENESISで実現
GENESISとは
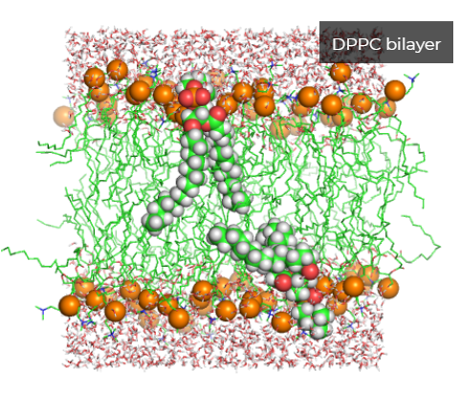
脂質二重膜
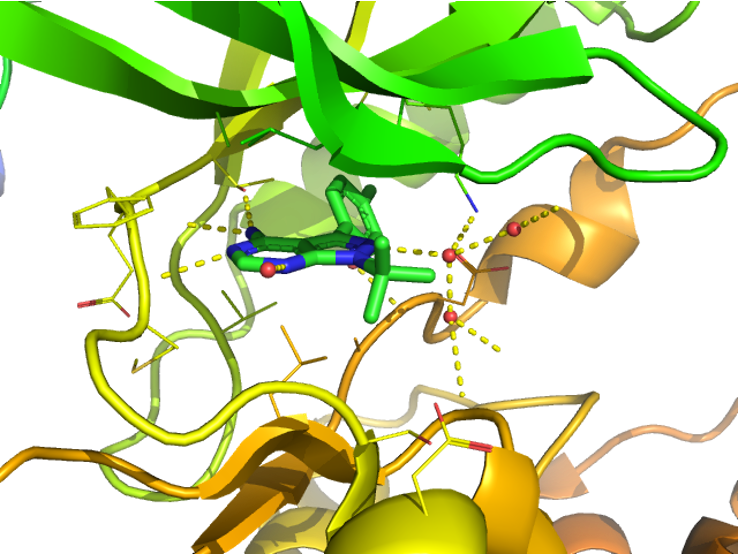
タンパク質と
リガンドの結合
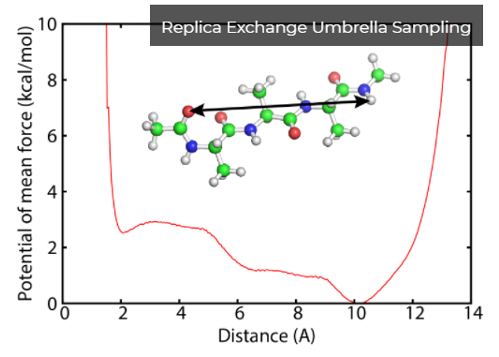
分子構造
自由エネルギー評価
GENESISは理化学研究所を中心に開発している分子動力学ソフトウェアで、生体分子を含む様々な分子システムのダイナミクスを計算します。
生命科学では、タンパク質や核酸(DNAやRNA)などの生体高分子が細胞という環境の中でどのように機能しているかを理解することが重要です。これまでに世界中でさまざまな分子動力学シミュレーションソフトウェアが開発されてきましたが、その多くは、少数のタンパク質や核酸などをターゲットとした比較的小さな分子系を主なターゲットとしていました。従って、多くのタンパク質や核酸、代謝物などで混み合っている細胞質などの細胞環境を考慮した分子動力学計算を効率的に行うことのできるソフトウェアはあまり多くありませんでした。
GENESISは、その開発当初から数千万から数億原子を含む巨大な生体分子系のダイナミクスを計算するために開発されています。そのため、「京」や「富岳」などのスーパーコンピュータのアーキテクチャ(基本設計)を考慮に入れた独自の計算アルゴリズムを導入しました。これによって、並列計算を最大限に高効率化することができ、「富岳」を用いることで最大10億個の原子を含む分子混雑環境のシミュレーションにも成功しています。
さらにGENESISは、レプリカ交換分子動力学法などの効率の良い構造探索アルゴリズムを複数備えており、タンパク質折れ畳みや凝集などの分子機構やタンパク質と薬剤の結合や相互作用などを解析することができます。また、新しいバージョンでは、原子一つ一つを粒子として考慮する全原子モデルだけでなく、複数の原子を束ねた粗視化粒子を用いた粗視化分子モデルとタンパク質の活性部位などの電子状態をあらわに考慮するQM/MMモデルを用いたシミュレーションを行うことができます。また、スーパーコンピュータのみならず、GPUワークステーション等でも利用することが可能です。このような手法は、生命科学や創薬などに応用できるだけでなく、高分子材料など様々な材料開発にも広く応用可能です。大学や研究機関などのアカデミックでの研究だけでなく、産業界などにも幅広く利用していただきたいと考えています。
GENESISの機能と特徴
GENESISとは何ですか?
- 「GENeralized-Ensemble Simulation System」は主に理化学研究所の杉田グループ(計算科学研究センター粒子系生物物理研究チーム、開拓研究本部杉田理論分子科学研究室、生命機能科学研究センター分子機能シミュレーション研究チーム)を中心に開発しています。
- タンパク質、脂質、核酸、糖鎖、およびそれらの複合体など様々な生体分子の分子動力学シミュレーションを行います。生体分子以外にも高分子材料など様々な分子系のダイナミクスを計算できます。
- フリーソフトウェア(LGPLv3)として配布されています。 (バージョン1.4またはそれより前のバージョンはGPLv2にて配布されています。)
GENESISの機能
- 独自の高速化機能、大規模な並列計算、GPUに対応し、「富岳」にも最適化されている。
- 全原子、粗視化、QM/MM計算が可能。
- RMSD(立体構造比較)やPCA(主成分解析による自由エネルギー地形)など、多くの解析ツールを含む
- レプリカ交換分子動力学法などの拡張アンサンブル法を用いた効率的な構造探索手法と自由エネルギー摂動法による水和や結合親和性の評価を実現
- タンパク質とリガンドの結合ポーズと親和性の予測
- 薬剤の溶解性などの予測
- 様々な計算ソフトウェア、モデリングツールや可視化ソフトウェア等と併用できる。
- CHARMM-GUIで作成した分子動力学や自由エネルギー計算の入力ファイルを直接GENESISで利用できる。
- VMDなどで、GENESISを用いたシミュレーション結果を可視化。
- QM/MM計算では、Gaussian, Q-chem, Tera-chem, Q-simulateなどの量子化学計算ソフトウェアを利用。
- J-OCTAからのインターフェイスを提供予定(J-OCTAでモデリング、結果解析)
GENESISのホームページ
以下のホームページから、ソフトウェアダウンロード、チュートリアル、講義資料、ベンチマークなどが利用可能です。
https://mdgenesis.org
GENESISのチュートリアル動画
本チュートリアルでは、水溶性タンパク質であるBPTI(牛の膵臓トリプシン阻害酵素)を例にとりGENESISで分子動力学法計算を行う方法を説明します。
GENESISのトピック
| 2024年1月18日 | ウイルスのスパイクタンパク質でウミホタルの発光基質が発光 |
|---|---|
| 2023年3月3日 | タンパク質の凝縮を制御する分子機構 |
| 2022年12月7日 | 膜ポンプによる膜輸送機構の普遍的概念の提唱 |
| 2022年4月25日 | スパイクタンパク質の構造変化を予測 |
| 2021年9月28日 | 生体膜カルシウムイオン輸送の分子機構を解明 |
| 2021年10月12日 | 国産の分子動力学ソフトウェア「GENESIS」の産業界における利用をサポートするサービスの提供開始 |
| 2021年8月19日 | 天然物が持つ鏡像異性な環状骨格を作り分ける |
| 2021年7月9日 | 混雑した細胞内で薬はどう効くのか |
| 2021年2月18日 | 新型コロナウイルス感染の分子機構を解明 |
| 2019年9月6日 | 酵素-阻害剤結合の初期会合体を予測 |
| 2018年10月19日 | クライオ電顕像からのタンパク質構造モデリングを高速化 |
| 2018年5月15日 | 1分子計測のデータ同化による生体分子構造ダイナミクス |
| 2018年2月5日 | 糖鎖構造から衝突断面積を予測 |
| 2017年8月29日 | 合成酵素と分解酵素の協演 |
| 2016年11月1日 | バクテリア細胞質の全原子分子動力学計算 |
| 2015年5月8日 | 超並列分子動力学計算ソフトウェア「GENESIS」を開発 |
GENESISの応用事例
マルチリゾレーション手法による細胞スケールシミュレーション
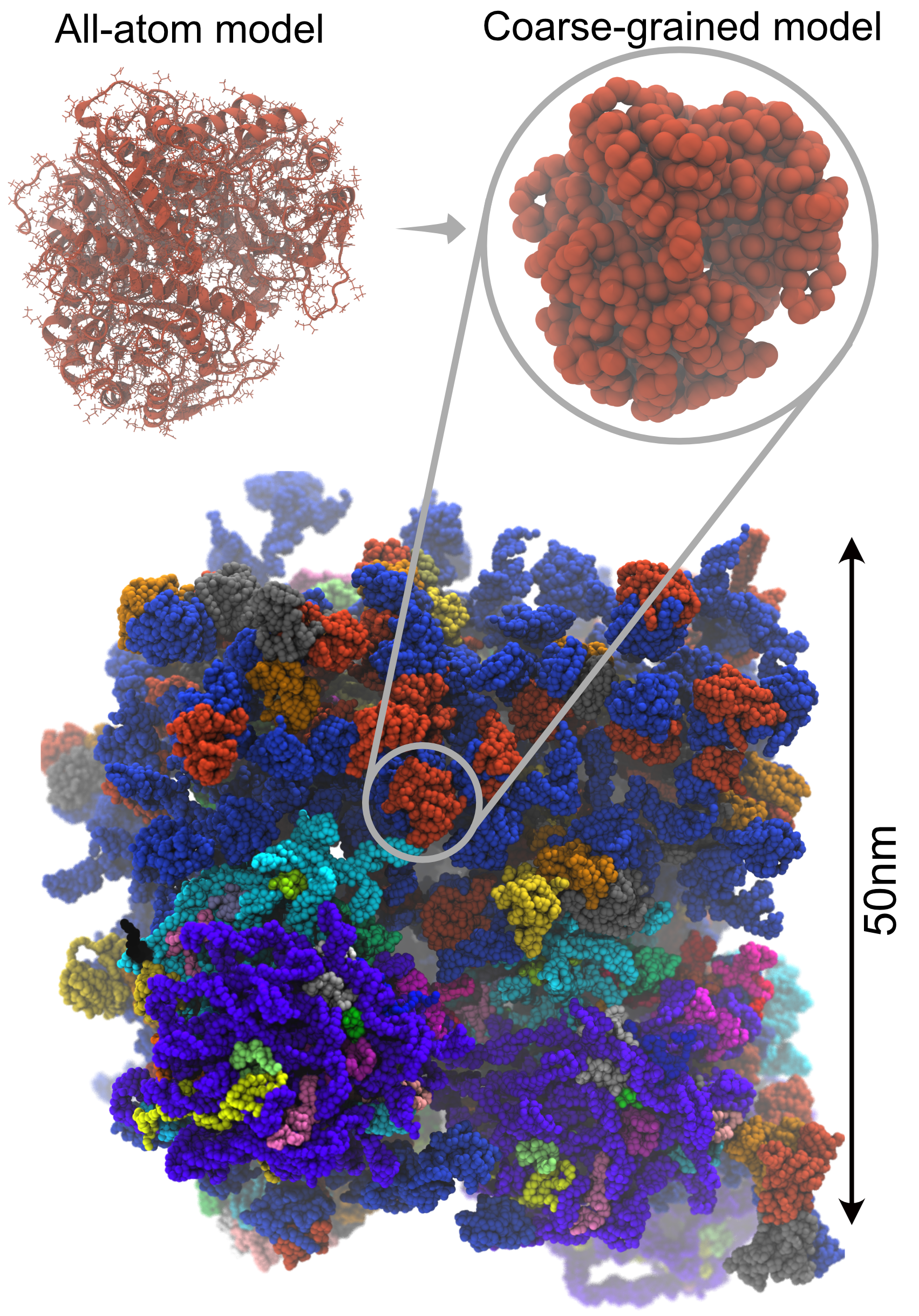
細胞内にはタンパク質、核酸、脂質などの様々な高分子が高濃度で含まれており、互いに相互作用することで機能を発現しています。このように多くの分子で混雑した環境内で、個々のタンパク質に特有な機能が正確に働いていることは決して自明なことではありません。ターゲットとするタンパク質同士、あるいはタンパク質と薬剤が、その他の多くの生体分子の存在下でどのようにして、互いを認識し、強く結合し、化学反応を生じさせているのでしょうか。このような問いに答える分子動力学シミュレーションを行うためには膨大な計算量が必要となり、従来は不可能でした。私たちは、GENESISを並列化・高度化することで、シミュレーションで観測可能な時間・空間的な範囲を広げるだけでなく、様々な粗視化分子モデルを開発・導入することで細胞内環境を考慮した大規模な生体分子系のシミュレーションを実現しました。さらに、個々の生体分子の機能、すなわち分子の結合や化学反応、を予測するために必要な自由エネルギー計算法やQM/MM計算法を開発しています。これらの多くの機能を用いたマルチレゾリューション手法を用いることで細胞スケールの生命現象の理解と予測を行います。
タンパク質と薬剤候補化合物の相互作用の予測
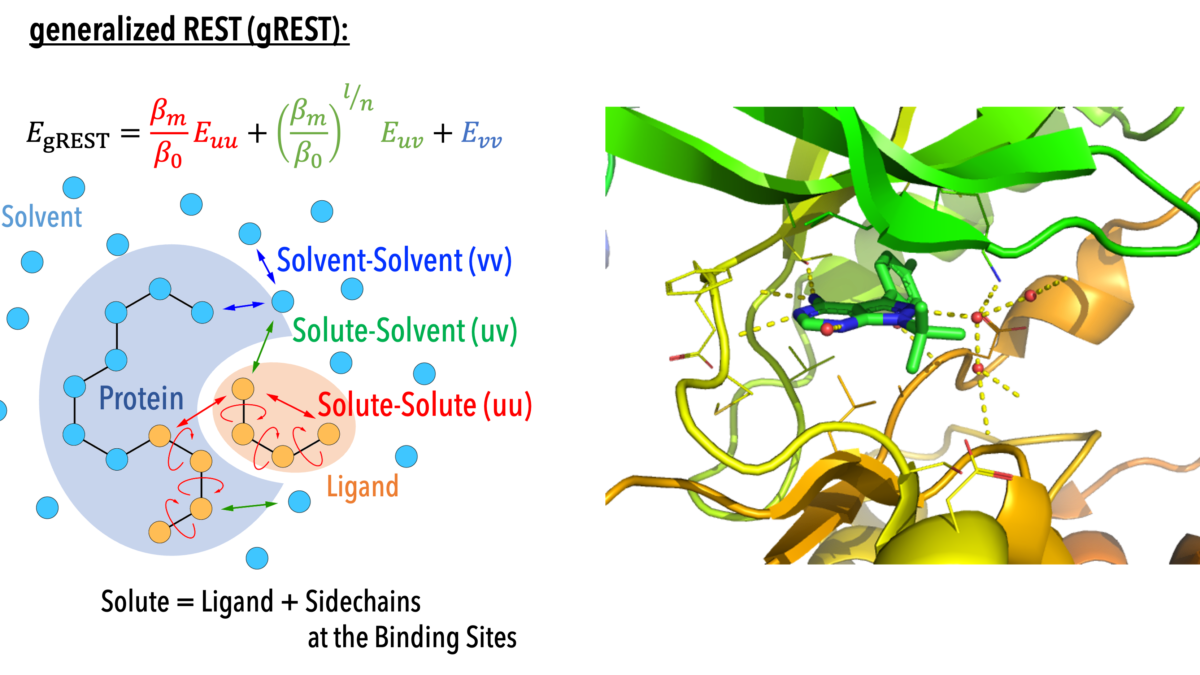
タンパク質がその機能を発現するために、基質と呼ばれる低分子あるいは別のタンパク質と結合し、様々な化学反応あるいは酵素反応を経て、細胞内での信号が伝達されていきます。細胞内で生じる信号伝達あるいは物質輸送は大変複雑で多くの生体分子が関与する非常に複雑なネットワーク構造を有しています。がんなどの疾患はこのようなネットワーク構造の破れに起因することがわかっていますが、疾患を治療するためにはタンパク質の機能を制御するための薬剤開発が必要です。計算機を用いたシミュレーションでは、疾患の原因因子と考えられるタンパク質をターゲットとして、そこに結合する薬剤候補化合物との相互作用、すなわち、タンパク質と薬剤候補化合物の複合体の立体構造やその結合親和性などを予測することができます。このような結合プロセスは、実際には長時間を有するレアイヴェントであることが知られているため、レプリカ交換分子動力学法などの効率的な構造探索手法、自由エネルギー摂動法などの計算手法などを組み合わせて、タンパク質と薬剤候補化合物の相互作用を評価することができます。私たちは、GENESISに導入した新しいアルゴリズムの効率を評価し、インシリコ創薬に応用できる新しい方法を開拓してきました。
実験データを統合した新しい動的構造生物学
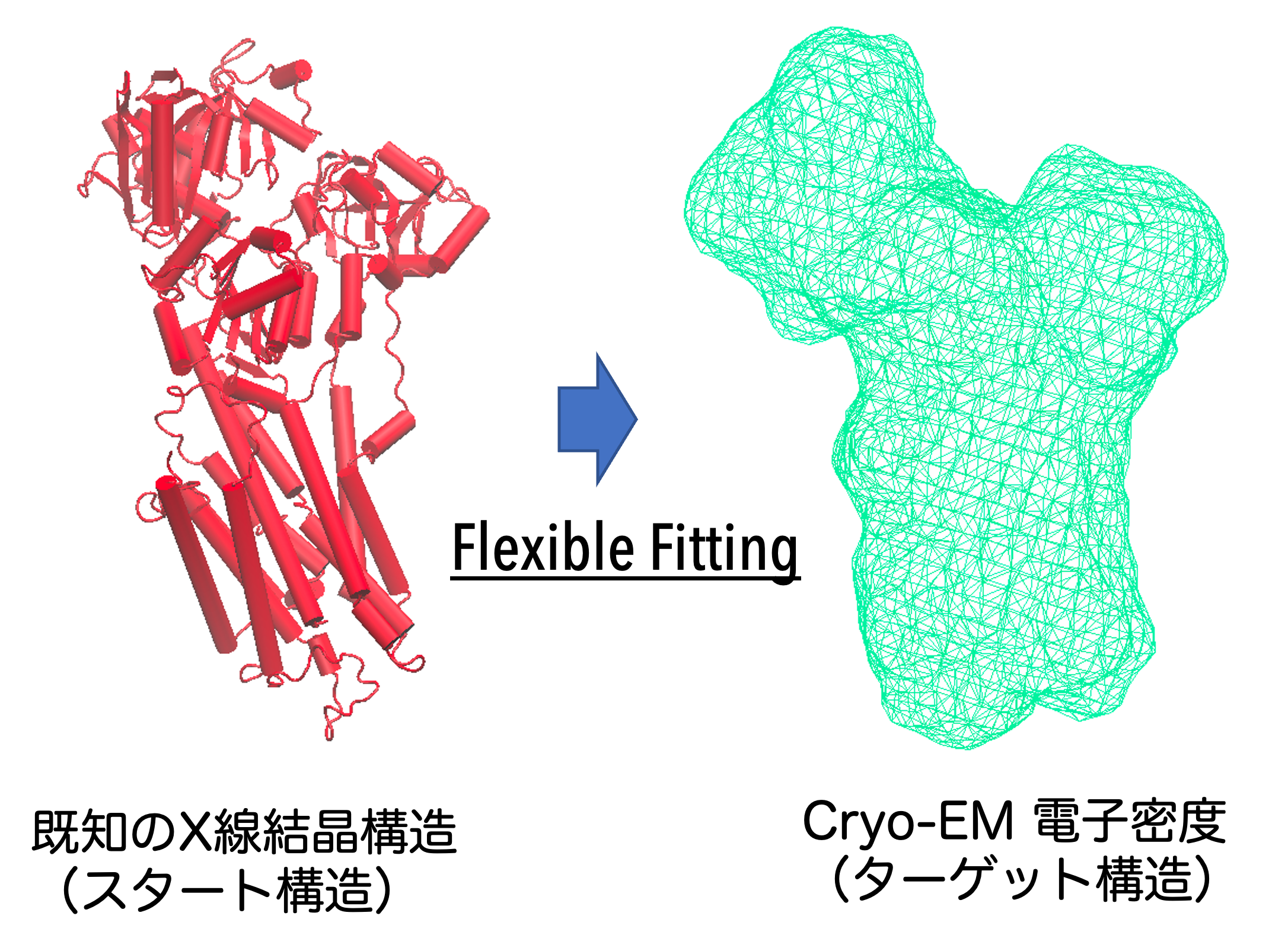
近年の構造生物学における技術の発展により、従来は原子構造を決定するのが困難であった生体分子複合体などの立体構造も短期間で決定できるようになりました。特に、クライオ電子顕微鏡を用いた単粒子解析技術を用いることで、タンパク質やその複合体の複数の立体構造を1回の実験で得ることも可能です。実験的に解かれた立体構造の解像度が向上している一方で、比較的柔らかい部分の構造や側鎖構造などの詳細が実験だけでは決定できないこともあります。このような問題を解決するために、クライオ電子顕微鏡で得られた電子密度などの実験データをシミュレーションに繰り込んだハイブリッド計算を行うことで、より信頼性の高い生体分子構造をモデリングすることができます。今後は、単粒子解析のみならず、核磁気共鳴(NMR)や分子分光学、1分子計測など様々な計測技術とシミュレーション研究の融合を目指しています。
新型コロナウイルス感染の分子機構を解明-「富岳」による新しい知見-
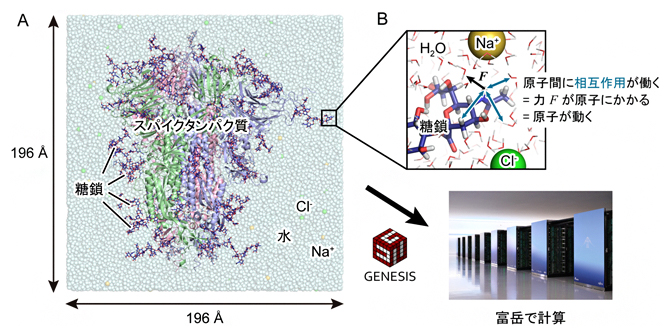
スーパーコンピュータ「富岳」と「Oakforest-PACS」を用いて新型コロナウイルスSARS-CoV-2の表面に存在する「スパイクタンパク質」のシミュレーションを行い、ウイルスがヒト細胞に侵入する際に起こるスパイクタンパク質の構造変化において、スパイクタンパク質表面を修飾している糖鎖が重要な役割を果たしていることを発見しました。
本研究成果は、新型コロナウイルス感染症 COVID-19に対する感染予防や治療に向けた医薬品の分子設計に貢献すると期待できます。
新型コロナウイルスのスパイクタンパク質の受容体結合ドメイン(RBD)には、「ダウン型構造」と「アップ型構造」が存在し、RBDがヒト細胞表面のACE2受容体に結合して感染する際はアップ型構造をとっていることが知られています。スパイクタンパク質の分子動力学シミュレーションを行いました。その結果、ダウン型構造とアップ型構造の両方において、スパイクタンパク質の表面を修飾している糖鎖が”補強役”となってRBDを安定化していることを発見し、RBD間の静電的な反発が駆動力となり、アップ型への構造変化が誘起されるという分子メカニズムを提案しました。
GENESIS on AWSの商用利用サポート
GENESIS on AWS
「富岳」の利用は「特定先端大型研究施設の共用の促進に関する法律」に基づき、利用促進業務を行う機関によって利用者選定の審査や報告書の提出が求められ、申請からアカウント発行までに、課題の種類によって数日から3カ月半程度の期間を要します。「GENESIS」を利用する企業にとっては、その利用目的が機密情報に当たるものが多く、またスピードも求められるため、特に企業競争力に直結するような秘匿性の高い開発において「富岳」利用には一部課題がありました。
こうした中、国立研究開発法人理化学研究所 計算科学研究センター(以下、R-CCS)は、「富岳」上で開発してきた研究成果であるシステムソフトウェアやアプリケーションを、クラウドサービスや他のスーパーコンピュータで誰もが直接利用できる環境を構築するといったクラウドの富岳化、いわゆる「バーチャル富岳」の実現に向けて取り組まれています。これにより、より多くの企業・研究機関に対して「富岳」を利用しやすい環境を整え、「富岳」ユーザーの拡大、技術の普及拡大、さらにこれらによるSociety 5.0(※1)の実現を目指されています。
「GENESIS on AWS」は、「バーチャル富岳」の提供に先行して、R-CCSが開発した分子動力学ソフトウェア「GENESIS」のAWS上での稼働を実現したものです。R-CCSでは、高い秘匿性とスピードが求められる創薬・材料開発分野に対して、「バーチャル富岳」に先駆けて提供することは意義深いと判断し先行提供を決定、理研数理が、この「GENESIS on AWS」の商用利用をサポートしていきます。
理研数理は、企業・研究機関からのお申し込みから最短1週間でAWSインスタンスを提供するほか、操作方法についても併せてサポートします。お客さまは「GENESIS」をタイムリーに利用することが可能となるほか、利用内容を公開する必要がないため、スピードと高い秘匿性が求められる研究・開発業務において、これまでよりさらに利用しやすくなります。加えて、理研数理は理化学研究所をはじめとするアカデミアと長年にわたる信頼関係を有しており、お客さまの研究・開発業務の高度化に向けて、研究者などによる技術指導や共同研究をコーディネートすることも可能です。
※1 「Society 5.0」とは、サイバー空間(仮想空間)とフィジカル空間(現実空間)を高度に融合させたシステムにより、経済発展と社会的課題の解決を両立する、人間中心の社会のことで、2016年に政府が「第5期科学技術基本計画」において日本が目指すべき未来社会の姿として初めて提唱されたもの。
GENESIS on AWSの商用サービスの特長
GENESISをAWS上で「富岳」上と同様に利活用可能
- 企業・研究機関が「富岳」上で創薬・材料開発の研究を実施し、秘匿性の高い開発時に制限の無いAWSに移行することが可能
- GENESISをAWS上で試用し、ステップアップで「富岳」に移行することが可能
- Fugaku OnDemand(統一的GUI環境)により、「富岳」もAWSも同じ使い勝手を実現
必要な時に、必要な量だけ、利用可能
- 理研数理が企業・研究機関からの依頼を受けて、最短1週間後にAWSインスタンスを提供
- 使用した計算資源量に応じた従量課金制
- 利用報告書は不要
GENESIS・AWSのインストール・環境構築・サポートがセットで提供される
- GENESIS、Fugaku OnDemandがインストール済
- 環境構築された、AWSインスタンスを提供
- GENESISとAWSの操作に関するサポートを理研数理がセットで提供
- ハイスキルのユーザーは、OSSのGENESISを自身でインストール・環境構築、計算資源を自身でAWSから直接購入した上で、AWS上でGENESISを利用することも可能
- 親会社のJSOLと協業し、必要なプリポスト機能の開発・提供を検討中
企業課題解決のための提案も受けられる
- 難易度の高い企業課題の解決や、研究・開発業務の高度化に向けて、R-CCS杉田先生と連携して、最適な解決策(技術指導、共同研究、等)を提案
- GENESIS Users’ Groupを通じて、GENESISの最新機能、利活用事例をタイムリーに共有
J-OCTAによるGENESISのモデリング
J-OCTA(ジェイ・オクタ)は、(株)JSOLが開発・販売している、材料・生命科学のためのシミュレーションソフトウェアで、2000年頃に国家プロジェクトで開発されたOCTA(オクタ)の商用版となります。国内外のメーカーや研究機関などで、多くの実績があります。最近ではデータサイエンス分野においても使われています。
J-OCTAは、ナノ~マイクロメートル(nm~μm)の マルチスケール・シミュレーションのためのモデリング機能を有しています。一般的に「分子シミュレーション」と呼ばれる分野になりますが、J-OCTAはその枠にとらわれず連続体分野のシミュレーション(流体・構造解析)も可能で、これにより幅広い現象に対応することができます。
J-OCTAの機能の一つとして、「全原子分子動力学(Full-Atomistic Molecular Dynamics : FAMD)」のモデリング機能があります。2次元の化学式からの3次元構造の構築や、PDBファイルのインポート、Amberなどの力場パラメータの設定などが可能です。特に生体分子のモデリングを目的として、「GENESISモデラ」のご提供を開始しています。GENESISの入力ファイルを作成して計算を実行し、結果ファイルをJ-OCTA上で可視化したり、様々な解析を実施することができるようになります。さらにJ-OCTAにはWindows版GENESISも同梱されていますので、すぐに計算を実行することが可能です。
図:GENESISモデラによるタンパク質のモデリング
先述の通り、J-OCTAにはマルチスケール・シミュレーションの機能が含まれます。例えば、粗視化モデルを用いることで、全原子分子動力学法では扱えないような大きなスケール(ポリマーの運動や相分離、両親媒性分子による二重膜/ミセル/ベシクルの形成など)、長時間の現象のシミュレーションが可能です。粗視化分子動力学(Coarse Grained Molecular Dynamics : CGMD)や、散逸粒子動力学(Dissipative Particle Dynamics : DPD)、平均場法(Mean Field Method)などが相当します。J-OCTA上でGENESISを用いることで、これらの粗視化モデルとの連携が可能となり、粗視化モデルで求めた構造を基にGENESISの初期構造を構築することができるようになります。また、J-OCTAには量子化学の機能も含まれていますので、点電荷の値の決定や力場パラメータの補正などで活用することができます。
図:粗視化モデル(DPD)によるベシクル形成
詳細はJ-OCTAのWebサイトでご確認下さい
GENESIS Users’ Group
GENESIS Users’ GroupはGENESISを利用している、またはその利用を検討している企業ユーザーと開発者が集うユーザーグループであり、GENESISの機能の開発とユーザ拡大・利用拡大を推進します。
次回開催予定の研究会はこちら
理化学研究所杉田グループのご紹介
理化学研究所杉田グループは、計算科学研究センター粒子系生物物理研究チーム、生命機能科学研究センター分子機能シミュレーション研究チーム、開拓研究本部杉田理論分子科学研究室で構成されています。この3つの研究チームに所属する研究員が、GENESIS (GENeralized-Ensemble SImulation System) に含まれる様々なアルゴリズムや計算手法を開発し、生体分子などの研究に応用しています。粒子系生物物理研究チームでは主に、GENESISの高度化・並列化と粗視化分子動力学法の開発を行っており、分子機能シミュレーション研究チームは様々な自由エネルギー計算法を開発し、タンパク質と薬剤候補化合物の相互作用の計算等に応用しています。杉田理論分子科学研究室ではさらに広い観点から分子科学と生物物理学を融合した新しい計算化学を開発すべく、QM/MM計算、データ駆動型シミュレーション、さらに機械学習等の情報科学を用いた研究を行っています。
計算科学研究センター(R-CCS) 粒子系生物物理研究チーム
https://www.r-ccs.riken.jp/research/labs/cbrt/
https://cbp.riken.jp/
生命機能科学研究センター(BDR) 分子機能シミュレーション研究チーム
https://www.bdr.riken.jp/jp/research/labs/sugita-y/index.html
https://bfs.riken.jp/
開拓研究本部(CPR) 杉田理論分子科学研究室
https://www.riken.jp/research/labs/chief/theor_mol_sci/index.html
https://tms.riken.jp/
